甲基化检测新方法—近红外荧光成像[创新技巧]
 2016-05-25来源:未知
2016-05-25来源:未知
现在发现了一种快速简便的基于NC膜的近红外荧光检测方法研究启动子CpG岛的DNA甲基化。在NC膜上设计捕获探针用于检测DNA甲基化以及由其引起的基因转录水平上的表达变化。这种方法不依赖于重亚硫酸盐处理和PCR扩增,具有更加宽广的动态范围以及相对较高的检测灵敏度。该新方法已经发表在《Analytical Biochemistry》杂志上。
作者研究目的为通过基于NC膜的近红外荧光成像方法检测启动子CpG岛DNA甲基化以及由此引起的转录水平上基因表达变化。文章中作者建立了两套检测体系:DNA甲基化体系和转录体系。检测原理如下图所示:
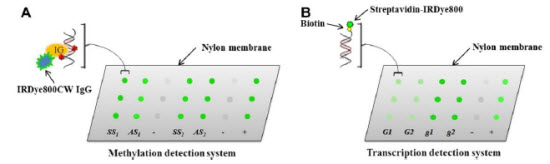
图A注解:甲基化检测体系。SS:正义链;AS:反义链。-:阴性对照,不含甲基胞苷的dsDNA。+:阳性对照,含甲基胞苷的dsDNA。IG:抗5-甲基胞嘧啶的抗体;IRDye800CW IgG:抗IgG的近红外二抗。
图B注解:转录检测体系。G1、G2为检测gene1(GAPDH)转录的两种探针;g1、g2为检测gene2(p14ARF)转录的两种探针。-:阴性对照(不含生物素的dsDNA)。+:阳性对照(含生物素的dsDNA)。
下面我们来看看具体的实验流程:
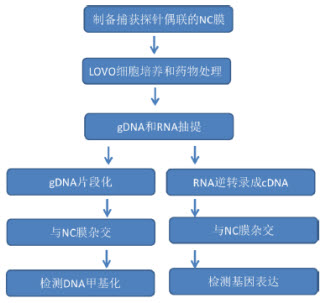
最终检测甲基化和基因表达时用的仪器为美国Licor公司的Odyssey近红外荧光成像系统。
认为基于近红外荧光成像的这种检测体系可以快速、高通量并且能准确定量DNA甲基化以及由其导致的转录水平上基因表达的变化。如下图实验结果:
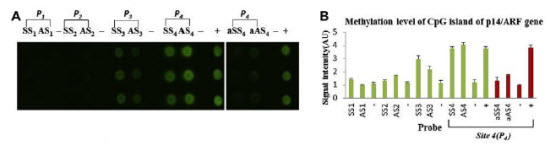
检测到肿瘤抑制基因p14ARF4个位点均有不同程度的甲基化。第四位点最强,第三位点中等强度,一、二位点最弱。当加入甲基化抑制剂5-aza-CdR以后,第四位点的甲基化减弱,再次证明了该结果的可靠性。
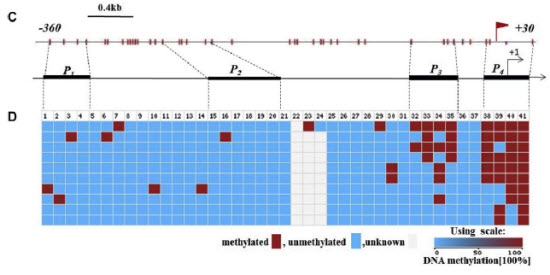
以上基于近红外检测体系的结果又同时用bisulfite sequencing的方法进行验证,结果如下图所示。与M-NIFA方法得到的结果一致。
同理,转录水平上基因表达也是同样的方法,Odyssey成像结果以及RT-PCR的验证结果如下图所示:药物处理后,肿瘤抑制基因p14ARF表达上调。RT-PCR与M-NIFA方法得到的结果一致。
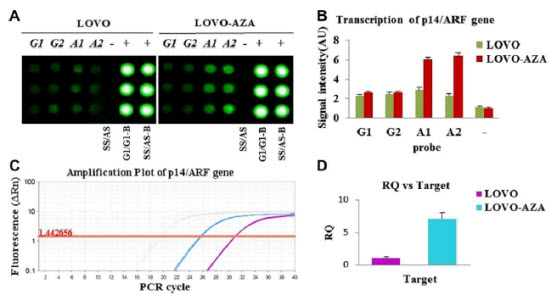
因此:基于NC膜的近红外荧光检测方法可用来检测启动子CpG岛的DNA甲基化以及由其引起的转录水平上基因表达。基于荧光的方法,可对DNA甲基化水平进行定量。不依赖于重亚硫酸盐处理,避免了耗时、对DNA有害、处理效率影响后续分析结果等的弊端,是一种快速有效的DNA甲基化检测方法。
此文转载来源:新浪博客



